Viele Fragestellungen in der modernen Biologie sind fast ausschließlich über experimentelle Ansätze zu beantworten, welche den Einsatz rekombinant klonierter Transkripte umfassen. Der Bereitstellung spezifischer Proteine mit maßgeschneiderten Linker- oder Markerfunktionen, so genannte Funktions-Tags, kommt dabei eine immer größere Bedeutung zu. Dies gilt insbesondere für die disziplinübergreifende Forschung in der Grenzflächen- und Nanotechnologie und der Molekularen Biologie, in der solche molekularen Werkzeuge eingesetzt werden.
Herstellung rekombinanter Proteine mit Funktions-Tag
Maßgeschneiderte Proteine für Proteomik-Anwendungen

Herstellung von Fusionsproteinen mit Funktions-Tags am Beispiel von EGFP (Enhanced green fluorescent protein) induziert in E. coli. Links: Durch IPTG (Isopropyl-beta-D-thiogalactosid) induziertes EGFP-Konstrukt in E.-coli-Pellets. Rechts: Als Kontrolle nicht-induziertes EGFP-Konstrukt.
Durch die Methoden der Gentechnik ist es möglich, nahezu jedes bekannte Gen aus einem beliebigen Organismus in ein geeignetes Expressionssystem einzubauen, so dass das Protein hergestellt und aufgearbeitet werden kann.
Das Fraunhofer IGB bietet das Design und die Herstellung solch maßgeschneiderter, biologisch aktiver Proteine im Milligramm-Maßstab als Forschungsdienstleistung an. Bei Interesse erstellen wir Ihnen gerne ein Angebot entsprechend Ihren Spezifikationen (Modifikation, Expressionssystem, Reinheitsgrad, Maßstab, Verwendungszweck).
Beispiele möglicher Anwendungen für Fusionsproteine:
- Protein/Protein-Interaktionen:
Identifizierung interagierender Proteine (Coimmunpräzipitationen, Two-Hybrid-Assays) - Darstellung von Proteinbiochips
- Gewinnung von Antikörpern gegen native Proteine
- Verwendung markierter Proteine (Fusionsproteine) zur Proteinlokalisation in mammalischen Zellen/Überexpression
- Anwendung von rekombinanten Peptiden und Proteinen in der klinischen Therapie
Klonierungs- und Expressionsstrategie
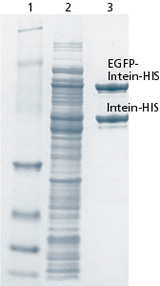
Bild 2: Überprüfung der Aufreinigung des hergestellten Fusionsproteins mit SDS PAGE. Spur 1: Proteingrößenmarker, Spur 2: Gesamtprotein nach Zellaufschluss (French Press), SDS-PAGE und anschließender Coomassie-Färbung aller Proteine. Spur 3: Aufgereinigtes Zielprotein (EGFP-Intein-HIS und Intein-HIS).
- Strategieplanung
- Datenbankrecherche
- Design des Konstruktes (Fusionsprotein,
Auswahl eines geeigneten Tags) - Auswahl eines geeigneten Expressionssystems
(eu- oder prokaryotisch) - Spezifisches Primerdesign (loops, hairpins)
- PCR-Amplifikation des »gene of interest«
- Enzymatische Manipulationen
- Ligation / Transformation in das entsprechende Klonierungssystem
- Screenen nach möglichen Mutanten
- Überprüfung der Klonierung über Sequenzierung
- Expression des Target-Proteins
- Induktionsoptimierung
- Zellaufschluss
- Verschiedene Aufreinigungsschritte
- Renaturierung
- Aktivitätsbestimmung
- Immobilisierung
- Messungen über MALDI-TOF-MS
Referenzen
Wir haben beispielsweise folgende Konstrukte erstellt und als native Proteine im Milligramm-Maßstab für die entsprechenden Anwendungen zur Verfügung gestellt:
- Das Cytokin Makrophagen-migrations-inhibierender Faktor (MIF) mit einem N-terminalen Streptactin-Tag (MIF-StrepTag)
- Enhanced green fluorescent protein / Intein /Chitin binding protein fusion protein (EGFP-Intein-CBD)
- Enhanced green fluorescent protein / Intein /10 x Histidine-tag fusion protein (EGFP-Intein-HIS)
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB