Flächendeckende Tests durch Hochdurchsatz-Diagnostik
Um flächendeckende PCR-basierte Tests anbieten zu können, muss der Durchsatz wesentlich erhöht werden. Im Rahmen des Vorhabens »Hochdurchsatz-Diagnostik von SARS-CoV-2« hat das IGB daher in Kooperation mit den Fraunhofer-Instituten für Zelltherapie und Immunologie IZI und für Produktionstechnik und Automatisierung IPA ein neues Verfahren zur Hochdurchsatz-Diagnostik von SARS-CoV-2 entwickelt und an klinischen Proben validiert.
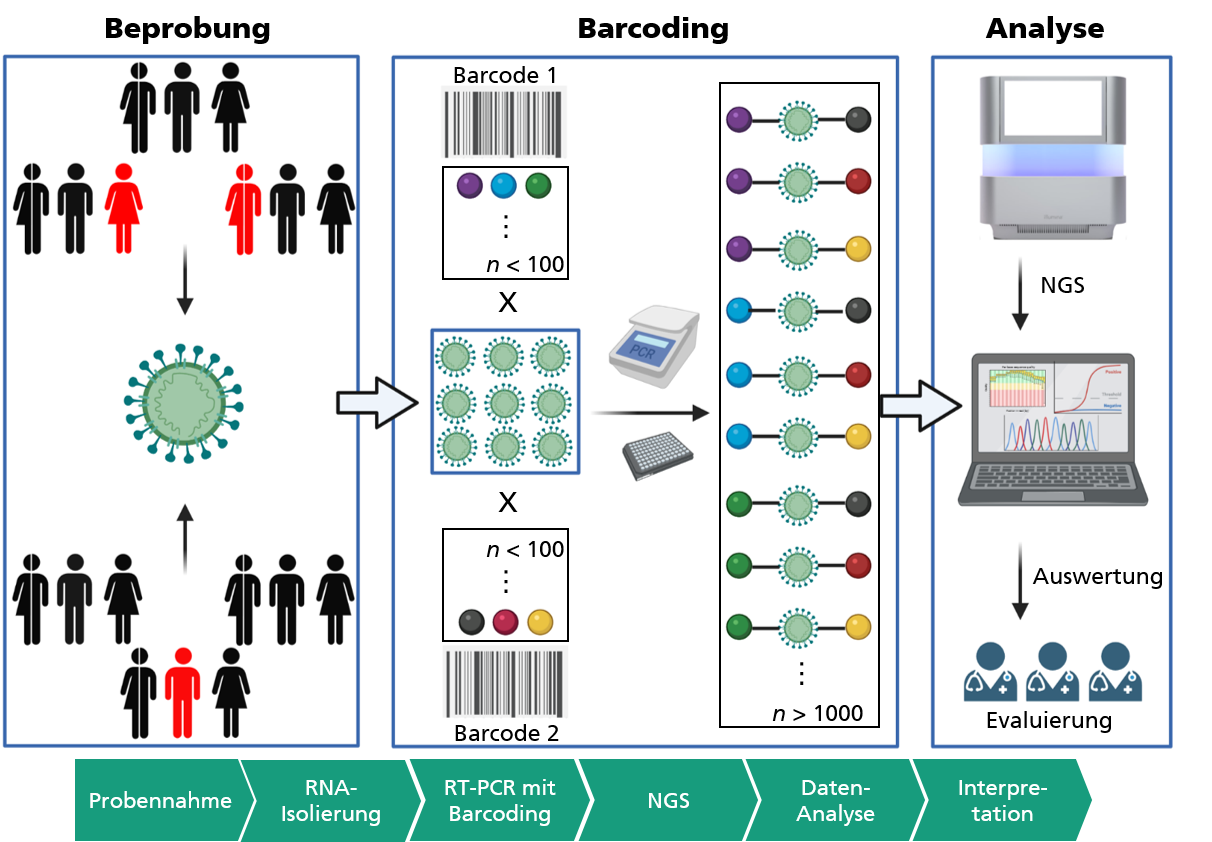
Dabei wird die reversen Transkription und Vermehrung spezifischer Virussequenzen (RT-PCR) mit der Hochdurchsatzfähigkeit moderner Sequenziertechnologien kombiniert. Um tausende Patientenproben gleichzeitig zu analysieren, werden diese in der RT-PCR mit spezifischen Oligonukleotiden – auch als »molekulare Barcodes« bezeichnet – gekennzeichnet, die eine eindeutige Zuordnung zur Probe ermöglichen (siehe Abb.).
Erste Validierung der Hochdurchsatz-Diagnostik
An insgesamt 672 klinischen Proben konnte CoV-2-KomET erfolgreich gestestet werden und erreichte dabei eine Spezifität von über 89 Prozent und eine Sensitivität von über 87 Prozent im Vergleich zur Standardbestimmung des PCR-Tests. Die diagnostische Güte von CoV-2-KomET, bestimmt durch die »Area under the curve (AUC)«, von 94 Prozent zeigt das Potenzial dieses Ansatzes, der zukünftig weiteren Evaluierungen und Verbesserungen unterzogen werden wird.
Neben der reinen SARS-CoV-2 Hochdurchsatz-Detektion konnte des Weiteren an 41 synthetischen Proben ein »respiratorisches Panel« erfolgreich etabliert werden und damit SARS-CoV-2 sowie Influenza A/B bis zu einer Nachweisgrenze von 10 Kopien/µL nachgewiesen werden.
Ausblick
CoV-2-KoMET als diagnostischer Ansatz, basierend auf der Hochdurchsatz-Sequenzierung von Virus-Nukleinsäuren, stellt eine vielversprechende und alternative Methode in Pandemien und bei Infektionsausbrüchen dar. Mit dem Ansatz konnten wir nicht nur den Probendurchsatz erhöhen, sondern durch die Skalierbarkeit auch die potenziellen Kosten pro Testung reduzieren. Darüber hinaus kann durch ein verändertes Design der spezifischen Virussequenzen innerhalb weniger Wochen auf neue Varianten reagiert werden.
Das Projekt wird mit dem industriellen Partner LMV Laboratories weitergeführt, mit einem besonderen Fokus auf der Verbesserung und Erweiterung des respiratorischen Panels, um Patienten möglichst simultan – mittels einer einzigen Probe – auf Infektionen mit SARS-CoV-2, Influenza und RSV zu testen.

 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB