Virulenzfaktoren bestimmen die Pathogenität
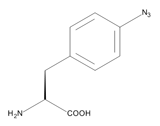
In den letzten Jahren ist die Anzahl an Erkrankungen durch humanpathogene Pilze stetig gestiegen. Aufgrund ihrer hohen Morbidität und Mortalität sind sie zu einem ernsten Problem im Gesundheitswesen geworden. Vor allem schwerwiegende Krankheitsverläufe wie systemische Mykosen, die den gesamten Körper betreffen, sowie entstehende Resistenzen bereiten Probleme in der Behandlung. Häufigster Auslöser systemischer Mykosen im Menschen ist der humanpathogene Pilz Candida albicans, der bei supprimiertem Immunstatus des Wirtes, beispielsweise in Folge von Operationen, Chemotherapien oder krankheitsbedingt, schwere Infektionsverläufe auslösen kann. Candida albicans verfügt über eine Vielzahl von Mechanismen, die zur Pathogenität führen. Vermittelt werden diese Mechanismen durch Virulenzfaktoren, Proteine mit unterschiedlichsten Funktionen in der Zelle. Virulenzfaktoren sind essenziell für die Pathogenität, insofern bieten sie ein vielversprechendes Ziel für die Entwicklung von Therapeutika. Voraussetzung dafür ist jedoch ein fundiertes Wissen über die molekularen Eigenschaften und physiologischen Interaktionsnetzwerke der Proteine in der Zelle. Da insbesondere für Candida albicans Techniken fehlen, um die Interaktionsnetzwerke von Proteinen in vivo zu analysieren, wurde am Fraunhofer IGB eine neue Methodik zur Analyse von Protein-Protein-Interaktionen mithilfe synthetischer Aminosäuren entwickelt.
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB