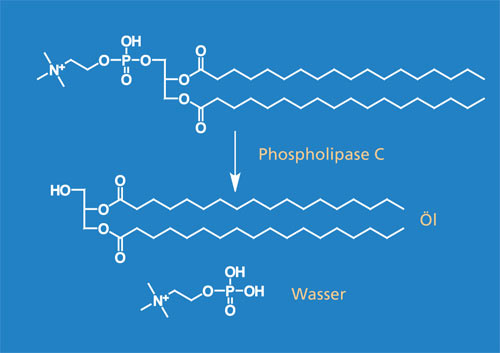
Enzyme finden bereits breite Anwendung in der Lebensmittel-, der Textil-, der Reinigungs- und Waschmittelindustrie, der Chemie- sowie in der Pharmaindustrie. In der Forschung werden durch moderne Methoden ständig weitere, neue industrierelevante Enzyme entdeckt, wissenschaftlich charakterisiert und hinsichtlich ihrer katalytischen Aktivität optimiert. Es wird geschätzt, dass in der Natur 10 000 Enzyme vorkommen. Davon sind 3000 bekannt, aber nur rund 120 Enzyme werden industriell genutzt [1].
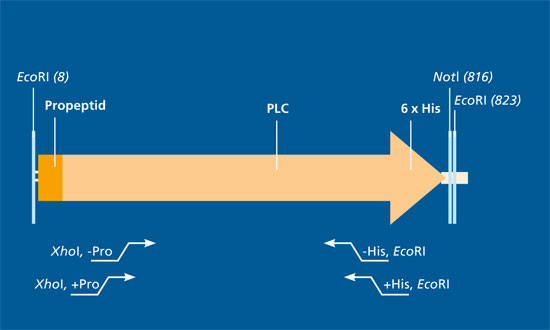
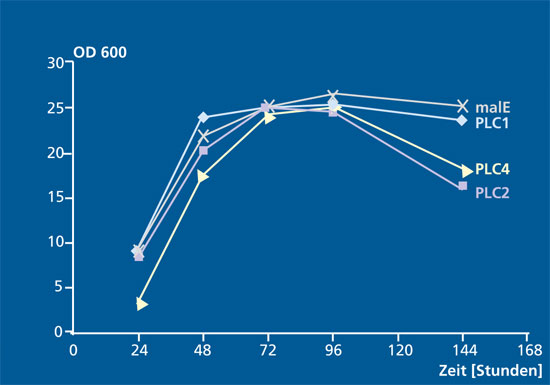
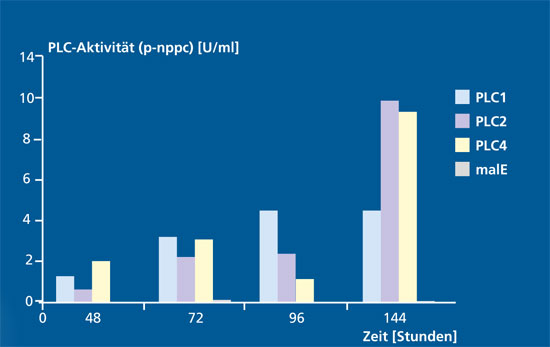
Das Fraunhofer IGB arbeitet daher zusammen mit Partnern aus Forschung und Industrie im Verbundprojekt »Innozym« mit dem Ziel, effiziente, rekombinante Expressionsverfahren zur Produktion technischer Enzyme zu entwickeln und diese bis zu einem Maßstab von 10 m3 herzustellen und aufzureinigen. Das Ziel soll durch die Entwicklung geeigneter Produktionsstämme und durch die Optimierung der Bioprozesstechnik erreicht werden.
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB