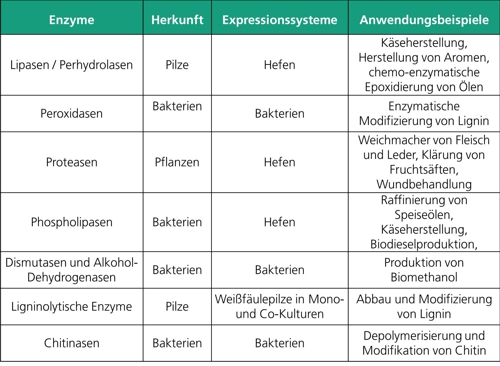
Für die Herstellung von Enzymen existieren verschiedene Verfahren. Eine Möglichkeit ist der direkte Einsatz von mikrobiellen Isolaten zur Enzymproduktion wie zum Beispiel Weißfäulepilze zur Herstellung ligninolytischer Enzyme oder chitinolytischer Bakterien zur Herstellung von Chitinasen. Viele dieser Mikroorganismen bilden bereits unter den richtigen Kultivierungsbedingungen und ohne gentechnische Modifikationen die gewünschten Enzyme in ausreichender Menge, die direkt oder nach Aufreinigung in industriellen Prozessen einsetzbar sind.
Oftmals produzieren Mikroorganismen jedoch nicht nur das gewünschte Enzym, sondern einen Cocktail an verschiedenen Enzymen. Viele mikrobielle Isolate sind zudem nicht für gentechnische Methoden zugänglich. Dadurch können kaum Optimierungen am Organismus vorgenommen werden, die beispielsweise die Enzymproduktion durch die Überexpression von Faltungshelfern verbessern können. Auch Modifikationen am Enzym sind nur schwierig durchzuführen, die die Aktivität, Spezifität oder Aufreingung optimieren würden (z. B. durch Veränderungen am aktiven Zentrum, oder durch das Anhängen von Affinitäts-Tags). In solchen Fällen kann das gewünschte Enzym heterolog in etablierten, mikrobiellen Produktionsstämmen wie Escherichia coli hergestellt werden.
Die Verfahrensoptimierung zur effizienten Herstellung einiger der genannten Enzyme wurde bereits durchgeführt. Die erzeugte Lipase wurde erfolgreich zur chemo-enzymatischen Epoxierung von Pflanzenölen eingesetzt.
Das Fraunhofer IGB beschäftigt sich sowohl mit der Identifizierung von mikrobiellen Isolaten, die gewünschte Enzyme oder Enzymgemische erzeugen, wie auch mit der Erzeugung von mikrobiellen Produktionsstämmen zur heterolgoen Enzymproduktion. Für die heterologe Enzymproduktion werden prokaryotische Expressionssysteme wie Escherichia coli oder Bacillus subtilis und eukaryotische Systeme wie die Hefe Kluyveromyces lactis und die methylotrophe Hefe Komagataella pastoris (früher Pichia pastoris ) eingesetzt.
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB