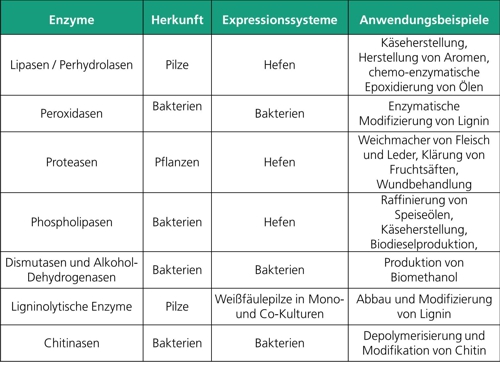
Das Fraunhofer IGB beschäftigt sich mit der Herstellung und Optimierung von Enzymen aus verschiedenen Enzymklassen, darunter vor allem hydrolytische Enzyme wie Lipasen, Phospholipasen und Proteasen, und Oxidoreduktasen wie Dismutasen, Dehydrogenasen und Peroxidasen.
Phospholipase C
Die Phospholipase C (PLC) katalysiert die Hydrolyse von Phospholipiden unter Bildung von Diacylglycerolen und wasserlöslichem Phosphorylethanolamin. PLC wird zur Raffination von Pflanzenölen großtechnisch eingesetzt, um eine schnellere und nahezu vollkommene Phasentrennung zu erreichen.
Lipasen
Lipasen nehmen aufgrund ihrer vielseitigen Einsatzmöglichkeiten ca. 5 % des Weltenzymmarktes ein. Lipasen katalysieren die Hydrolyse von Fetten (Triglyzeride) in Glyzerin und Fettsäuren. Je nach Reaktionsbedingungen können Lipasen weitere Reaktionen katalysieren, wie zum Beispiel Veresterungen, Umesterungen, Alkohollyse, Säurehydrolyse, Aminolyse, Acylierungen, Perhydrolysen und Epoxidierungen.
Proteasen
Proteasen (EC 3.4) gehören zu den wichtigsten Enzymen der Industrie. Ca. 60 Prozent aller weltweit genutzten kommerziellen Enzyme sind Proteasen. In der Industrie werden Proteasen vor allem in der Pharmazie, der Medizin und der Lebensmittelindustrie eingesetzt. Pflanzen bieten ein weites Spektrum verschiedenster Proteasen mit unterschiedlichsten Eigenschaften. Die Nutzung und Produktion pflanzlicher Proteasen für die Industrie ist jedoch, im Gegensatz zur Herstellung mikrobieller Proteasen, durch die oft niedrigen Ausbeuten und die schwierigen Extraktionsbedingungen eingeschränkt. Durch biotechnologische Verfahren und die rekombinante Herstellung pflanzlicher Enzyme in mikrobiellen Expressionssystemen, wie z. B. Hefen, können pflanzliche Enzyme in großen Mengen für die Industrie zugänglich gemacht werden.
Formaldehyd-Dismutase
Eine Formaldehyd-Dismutase aus Pseudomonas sp., die bereits seit einigen Jahren erfolgreich aus dem Wildtypstamm am Fraunhofer IGB erforscht wurde, besitzt zwar einen Cofaktor, aber auch einen integrierten Mechanismus zur Regenerierung desselbigen, sodass diese Oxidoreduktase gut für großtechnische Prozesse geeignet ist. Das Substrat Formaldehyd sowie die Produkte Methanol und Ameisensäure sind wichtige Plattformchemikalien, die in der chemischen Industrie in großen Mengen verarbeitet werden. Gegenwärtig beschäftigt sich das Fraunhofer IGB mit der heterologen Expression dieses Enzyms in Escherichia coli . Eine weitere Oxidoreduktase, die Methanol-Dehydrogenase, ist von industrieller Relevanz, da nur wenige Alkohol-Dehydrogenasen deutliche Aktivität gegenüber der Plattformchemikalie Methanol besitzen. Die Arbeiten am Fraunhofer IGB hierzu umfassen die heterologe Expression des Enzyms sowie dessen Immobilisierung und Stabilisierung im Hinblick auf den Einsatz in großtechnischen Prozessen. Gemeinsam mit der Formaldehyd-Dismutase soll dieses Enzym zur Herstellung von Methanol aus Biogas eingesetzt werden.
Peroxidasen
Peroxidasen, ebenfalls den Oxidoreduktasen zugehörig, werden am Fraunhofer IGB hinsichtlich ihrer ligninolytischen Aktivitäten untersucht. Neben Peroxidasen, die von Weißfäulepilzen zum Abbau von Lignin sekretiert werden, beschäftigt sich das Fraunhofer IGB mit neuen bakteriellen Peroxidasen und deren Rolle im Lignin-Abbauprozess. Die bakteriellen Dyp-type Peroxidasen sind eine eigene Klasse der Haem-Peroxidasen und kommen in Pilzen sowie Bakterien vor. Einige dieser Dyp-type Peroxidasen sind vermutlich am Abbau von Lignin beteiligt. Am Fraunhofer IGB werden bakterielle Dyp-type Peroxidasen zunächst im Labormaßstab heterolog in Escherichia coli exprimiert und die Enzymausbeute optimiert. Des Weiteren werden die Enzyme gereinigt und ihre katalytischen Eigenschaften untersucht. Eine spätere Anwendung kann im Bereich der Lignin-Modifizierung oder der Oxidation von aromatischen Substanzen bei der Abwasseraufbereitung liegen.
Chitinasen
Eine weitere Klasse industriell relevanter Enzyme, welche am Fraunhofer IGB hergestellt werden, umfasst die Chitinasen. Diese sollen zur Monomerisierung von Chitin eingesetzt werden, wodurch ein Wertstoffstrom aus dem Abfallstoff Chitin generiert werden soll (siehe Wertstoffherstellung aus Abfallströmen).
 Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB
Fraunhofer-Institut für Grenzflächen- und Bioverfahrenstechnik IGB